基于上一次的降维文件,我们可以查看一下其降维质量如何:
sc.pl.umap( adata, color=["total_counts", "pct_counts_mt", "scDblFinder_score", "scDblFinder_class"],)
在这里我们选择了四个指标用于生成可视化图像。
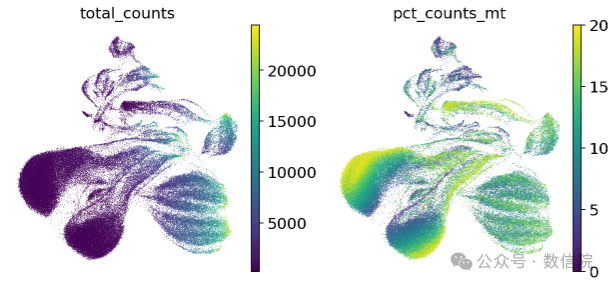
umap图1
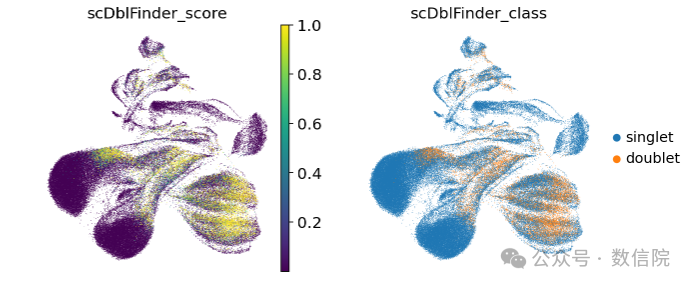
umap图2
现在我们将这个结果进行保存,并继续后续的分析。
在 scRNA-seq 数据分析中,我们在数据集中描述细胞结构,并找到与已知细胞状态或细胞周期阶段相关的细胞身份,这个过程通常称为 cell 身份注释。为此,我们将细胞构建成簇以推断相似细胞的身份。
聚类本身是一个常见的无监督机器学习问题。 我们可以通过最小化简化的表达式空间中的簇内距离来推导出簇。在这种情况下,表达空间决定了细胞相对于降维表示的基因表达相似性。例如,这种低维表示是通过主成分分析确定的,然后相似性评分基于欧几里得距离。
在 KNN 中,图形由反映数据集中单元格的节点组成。我们首先在所有细胞的 PC 简化表达空间上计算欧几里得距离矩阵,然后将每个细胞连接到其 K 个最相似的细胞。通常,K 设置为 5 到 100 之间的值,具体取决于数据集的大小。KNN 图通过将相对于表达空间的密集区域表示为图中的密集连接区域来反映表达数据的底层拓扑 。KNN 图中的密集区域通过 Leiden 和 Louvain 等检测方法检测。
Leiden是 Louvain的改进版本,其性能优于其他用于单细胞 RNA-seq 数据分析的聚类方法。由于 Louvain 算法不再维护,因此首选使用 Leiden。
因此,我们建议在单单元 k 最近邻 (KNN) 图上使用 Leiden 算法来对单单元数据集进行聚类。Leiden 通过考虑集群中单元之间的链接数与数据集中总体预期链接数来创建集群。
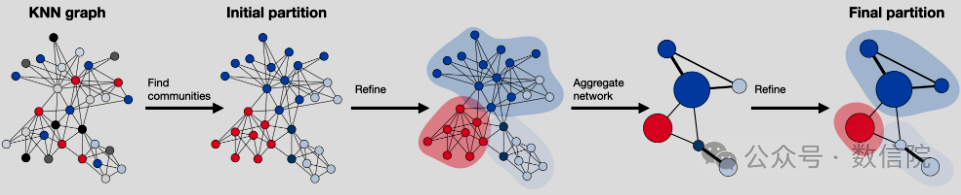
Leiden 算法计算从 PC 约化表达式空间获得的 KNN 图上的聚类。它从一个初始分区开始,其中每个节点都来自自己的社区。接下来,该算法将单个节点从一个部分移动到另一个集团以找到一个分区,然后对其进行优化。基于优化的分区,生成聚合网络,再次优化该网络,直到无法获得进一步的改进,并达到最终分区。
Leiden 模块有一个resolution参数,该参数允许确定分区集群的规模,从而确定集群的粗糙度。分辨率参数越高,聚类越多。该算法还允许通过对 KNN 图进行子集化,对数据集中的特定集群进行高效的子聚类。子聚类使用户能够识别聚类中的细胞类型特定状态或更精细的细胞类型标记,但也可能导致仅由于数据中存在的噪声而产生的模式。
这一步在scanpy中就可以进行,但由于我们刚才所保留的那个文件已经进行了降维分析,因此我们选用其之前的质控完成的文件。
import osos.environ["KMP_DUPLICATE_LIB_OK"]="TRUE"
我们先简单的在不运行Leiden算法的情况下umap可视化:
sc.pp.neighbors(adata, n_pcs=30)sc.tl.umap(adata)
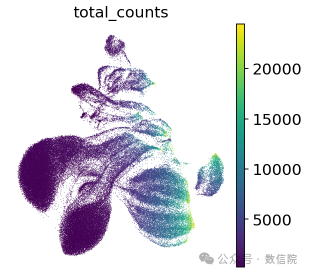
umap
scanpy 中的默认 resolution 参数是 1.0。但是,在许多情况下,分析人员可能希望尝试不同的分辨率参数来控制聚类的粗糙度。因此,我们建议将集群结果保存在指示所选分辨率的指定键下。现在,我们将使用 Leiden 算法在不同分辨率下获得的不同聚类结果可视化。分辨率在很大程度上影响了我们的聚类的粗糙程度。较高的分辨率参数会导致更多的群落,即更多的已识别集群,而较低的分辨率参数会导致更少的群落。因此,resolution 参数控制算法如何将 KNN 嵌入中密集聚集的区域组合在一起。这对于注释集群尤为重要。
sc.tl.leiden(adata, key_added="leiden_res0_25", resolution=0.25)sc.tl.leiden(adata, key_added="leiden_res0_5", resolution=0.5)sc.tl.leiden(adata, key_added="leiden_res1", resolution=1.0)
可以看到已经添加了层级了:
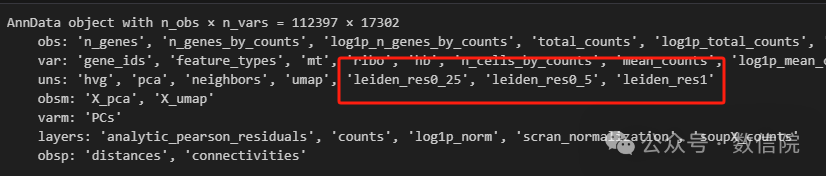
adata层级内容展示
接下来我们再可视化一下:
sc.pl.umap( adata, color=["leiden_res0_25", "leiden_res0_5", "leiden_res1"], legend_loc="on data",)
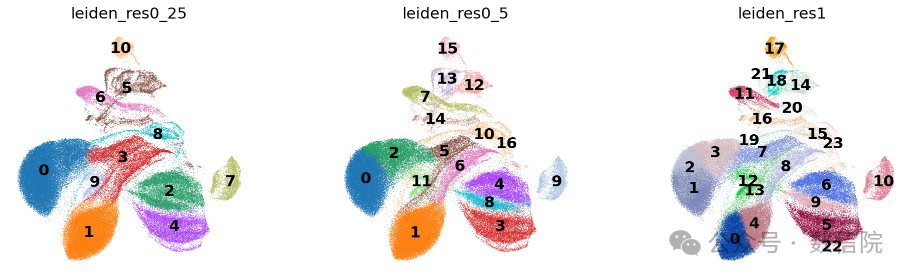
不同参数值umap图